Protein Co-Regulation Map Will Offer Better Insights on Uncharacterized Protein Functions. / La mappa di coregolamentazione delle proteine offrirà migliori approfondimenti sulle funzioni delle proteine non caratterizzate.
Protein Co-Regulation Map Will Offer Better Insights on Uncharacterized Protein Functions. The process of the ENEA RM2012A000637 patent is very useful in this application. / La mappa di coregolamentazione delle proteine offrirà migliori approfondimenti sulle funzioni delle proteine non caratterizzate. Il procedimento del brevetto ENEA RM2012A000637 è molto utile in questa applicazione.
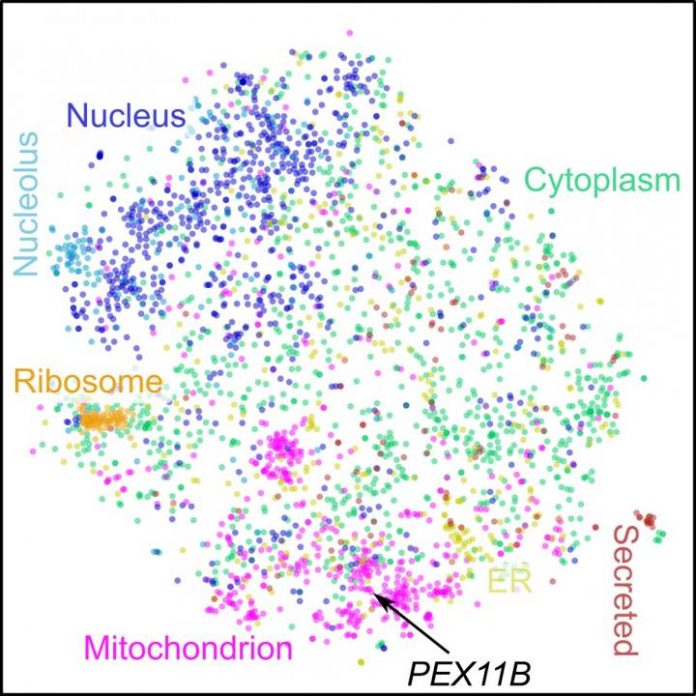
Scientists reported (“Co-regulation map of the human proteome enables identification of protein functions”) in Nature Biotechnology that they have produced a co-regulation map of the human proteome, which was able to capture relationships between proteins that do not physically interact or co-localize. This will enable the prediction and assignment of functions to uncharacterized human proteins, according to the researchers. The co-regulation map can be explored at www.proteomeHD.net.
Many of the functions of human proteins are still unknown, but researchers at the Wellcome Centre for Cell Biology, University of Edinburgh, and the Institute of Biotechnology, Technische Universität (TU) Berlin have applied large-scale quantitative proteomics and machine-learning to produce a protein covariation dataset of the human proteome. The dataset forms the basis of a co-regulation map which can be used to predict the potential function of uncharacterized human proteins.
“Assigning functions to the vast array of proteins present in eukaryotic cells remains challenging. To identify relationships between proteins, and thereby enable functional annotation of proteins, we determined changes in abundance of 10,323 human proteins in response to 294 biological perturbations using isotope-labeling mass spectrometry. We applied the machine learning algorithm treeClust to reveal functional associations between co-regulated human proteins from ProteomeHD, a compilation of our own data and datasets from the Proteomics Identifications database,” the investigators wrote.
“This produced a co-regulation map of the human proteome. Co-regulation was able to capture relationships between proteins that do not physically interact or colocalize. For example, co-regulation of the peroxisomal membrane protein PEX11β with mitochondrial respiration factors led us to discover an organelle interface between peroxisomes and mitochondria in mammalian cells. We also predicted the functions of microproteins that are difficult to study with traditional methods.”
“In this study, we took thousands of mass spectrometry experiments that other laboratories had published over the past few years and re-purposed them in a way that was completely unrelated to what the original authors had intended. We used a machine-learning algorithm to mine this huge collection of data and managed to assign a biological function to hundreds of proteins (genes) that were previously uncharacterized,” said Juri Rappsilber, PhD, Wellcome senior research fellow, professor of proteomics at the University of Edinburgh, and professor of bioanalytics at Tu Berlin, and Georg Kustatscher, PhD, a postdoctoral researcher at the Wellcome Trust Centre for Cell Biology.
Exploring the map revealed unexpected co-regulation partners, namely the peroxisomal membrane protein PEX11β with mitochondrial respiration factors. In cooperation with Michael Schrader, PhD, and his team at the University of Exeter, this led to the identification of a novel interaction between two crucial cellular organelles (mitochondria and peroxisomes).
“Peroxisomes and mitochondria in mammals are intimately linked and cooperate in the breakdown of fatty acids and cellular energy balance. Using live cell imaging we revealed that PEX11β mediates the formation of membrane protrusions, which emanate from peroxisomes and interact with mitochondria. They likely function in the metabolic cooperation and crosstalk between both organelles and may facilitate transfer of metabolites during mitochondrial energy (ATP) production,” said Schrader.
The scientists at the University of Edinburgh created the co-regulation map to allow users to search for a protein of interest, showing its position in the co-regulation map together with any co-regulation partners. They say the map is interactive and zoomable, making it easy to explore the neighborhood of a query protein. It is designed to support researchers in exploring co-regulation data at multiple scales, to validate existing hypotheses or to create new ones.
“With an ever increasing amount of protein expression data being made available, protein co-expression analysis has huge potential for gene function annotation. In a time when `big data’ becomes more and more relevant for life science, a key lesson we learned from this project is: never throw away your data—they can be re-purposed, recycled, and with the right tools there is plenty more information and knowledge that can be extracted from them,” noted Kustatscher.



Commenti
Posta un commento