Spatial Omics Spotlights the Players in the Tumor Microenvironment / L'omica spaziale mette in luce gli attori del microambiente tumorale
Spatial Omics Spotlights the Players in the Tumor Microenvironment. The process of the ENEARM2012A000637 patent is very useful in this type of application./ L'omica spaziale mette in luce gli attori del microambiente tumorale. Il procedimento del brevetto ENEARM2012A000637 è molto utile in questo tipo di applicazione.
Segnalato dal Dott. Giuseppe Cotellessa / Reported by Dr. Giuseppe Cotellessa
Researchers are using spatial omics to look deeper into the tumor microenvironment and unravel tumor heterogeneity with an eye on gleaning important clinical insights.
Many hard-to-treat cancers often recur months or years after successful treatment. “You could have one cell that escapes treatment and it's that one cell that will populate and be resistant and allow for a recurrence to happen,” said Jasmine Plummer, founding director of the Center for Spatial Omics at St. Jude Children's Research Hospital.
Investigating such tumors with even single-cell omics technologies could miss these resistant cells. Before analysis, single-cell technologies destroy the cancer tissue to look at what's happening in the tissue as a whole. However, a lot of the interesting stuff inside tumors happens at the level of individual cells and depends on the context in which they exist. Single-cell technologies lose this spatial context when the cells are broken up.
This is where spatial omics come in.
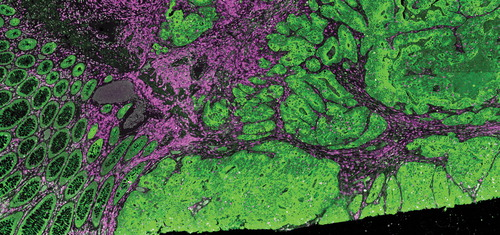
With advances in omics technologies, cancer biologists have extensive information on the genes, proteins, and other metabolites that make up the messy environment of a tumor. Single-cell omics goes further, enabling the identification of all cell types in a tumor sample. This has only deepened our understanding of the extreme heterogeneity of tumor cells. Spatial omics technologies are placing these insights in the spatial context.
Take gene expression, for example. Single-cell transcriptomics reveals which genes are being expressed across different cell types. But it doesn't say where these cells are in the tumor. Spatial transcriptomics technologies fill this gap by simultaneously recording spatial coordinates with gene expression data. This is the crux of the growing field of spatial omics: assigning pin codes to omics data.
Spatial transcriptomics technologies such as in situ hybridization and in situ sequencing allow researchers to capture transcriptomes without losing spatial information. The former uses fluorescent, gene-specific probes that bind mRNAs, whereas the latter sequences the transcripts directly in a section of a fixed tissue.
Complementing these imaging-based methods are other spatial technologies based on next-generation sequencing. These include high-definition spatial transcriptomics (HDST) and deterministic barcoding in tissue for spatial omics sequencing (DBiT-Seq). HDST uses spatially barcoded bead arrays to map RNA to location on histological sections. DBiT does the same for both proteins and RNA, enabling investigation of RNA-protein interactions with a spatial context. Technologies to study proteins and other metabolites are similar in principle to those developed to study transcripts. More recently, the push toward investigating multiple layers of information at once is driving the development of spatial multiomics tools like DBiT.
The Diverse Tumor Microenvironment
Cancer cells must continuously evade the immune system while establishing the infrastructure to support their uncontrolled growth. That infrastructure requires careful communication with a diverse coterie. It includes healthy cells in the host tissue, immune cells that must be tricked, and blood vessels that the tumor needs to spread, among others. These make up a dynamic ecosystem known as the tumor microenvironment (TME) which plays a critical role in the growth and spread of a tumor.
By integrating single-cell omics and spatial omics, researchers finally have a way to create detailed maps of different tissues, including cancers. In an article published in Molecular Systems Biology, researchers created a single-cell atlas of the human liver TME. The researchers found recurring interactions between the carcinoma cells and stromal cells around them, suggesting that drugs that target these interactions could be more broadly applicable.
In another study published in Cell, scientists showed that immune T cells exhibit a remarkable phenotypic diversity in the breast TME. Not only do they far outnumber the types of T cells in healthy breast tissues but also the activation states of the tumor T cell types are in a continuum. Further, the study suggested that the diversity of the immune cells was a consequence of the diversity of local microenvironments within the tumor.
Tumors have a complicated immune ecology that helps them “become invisible to the immune system or tell the immune system to stop sniffing so it doesn't find it or tell the T cells not to try to kill tumor cells,” said Joseph Lehar, senior vice president of R&D strategy at Owkin, a French-American AI biotech startup.
In tumor immune biology, the decisions made by cells about whether to kill cells suspected to be tumors are highly localized. “It's all about what a particular tumor cell or group of cells looks like and the immune cells are sensing that and then responding to it in a particular way,” Lehar added. A spatial understanding of the immune microenvironment of tumors is critical to be able to tackle untreatable cancers. This is because a tumor's ability to evade the immune system is central to both how it establishes the required architecture to grow and how it resists treatments.
Another critical aspect of the TME is the presence of a hypoxic niche. A phenomenon that marks malignant tumors, this is a less-than-physiological level of oxygen that underlies the different cell signaling architecture within tumors. In a study published in Immunity, researchers showed that hypoxic niches attract and hide tumor immune cells. Combining spatial information with single-cell transcriptomics, they demonstrated that crosstalk between glioblastoma and immune cells in the hypoxic niche is crucial in suppressing the immune system.
The TME also consists of additional mechanisms that shape the fate of a tumor by their interactions with both cancer and healthy cells. These include the extracellular matrix, free lipids, and mechanical cues, to name a few. To map these components along the tumor, researchers leverage other spatial profiling technologies, often used in conjunction with spatial omics, such as multiplexed imaging and mass cytometry.
In multiplexed imaging, “we use antibodies to detect a huge range of different proteins in the very same tissue section,” said Charlotte Stadler, codirector of the Spatial and Single Cell Biology platform and head of the spatial proteomics unit at the Swedish national research center, SciLifeLab. This allows researchers to do “deep phenotyping and know what cell types are close in space and potentially interacting with one another.” Deep phenotyping, a term increasingly in focus in the precision medicine space, refers to phenotyping at multiple levels of omics data.
Mass cytometry, similarly, allows researchers to track far more metabolites than conventional cytometry techniques. Such spatial profiling techniques are also critical to enable three-dimensional (3D) spatial omics. While maps of tumors obtained with spatial omics are often two dimensions (2D), tumors themselves exist in 3D. 3D spatial omics provide a complete picture of what's happening within tumors and, consequently, better insights.
Tumors Are Highly Heterogeneous
Different cells within a tumor have different genotypic and phenotypic makeup. Even in a genetically homozygous population of cancer cells within a tumor, the cells differ considerably phenotypically. Genomic instability, considered an important hallmark of tumor development, is the main driver of high cellular diversity in tumors.
The high diversity of cells within a tumor “is pivotal for comprehending the multifaceted functions of tumor cells and their intricate relationship with the microenvironment,” said Dana Adel Mustafa, a cancer researcher at the Erasmus University Medical Center.
By applying spatial omics techniques to cancer tissues, biologists are learning more about the heterogeneity of different tumors. Spatial omics technologies are rapidly advancing cancer research by producing hypothesis-generating data on tumor heterogeneity. For example, in a study published in Cancer Cell, researchers used spatial transcriptomics to investigate the heterogeneity in renal cell carcinoma. They observed that intratumoral heterogeneity far outweighs that of somatic mutations in kidney cancer.
More specifically, the location of immune T cells in the tissue, rather than the mutations they have accumulated, primarily defines the level of their dysfunction. This study was part of the Human Cell Atlas consortium, a wider effort to create a comprehensive atlas of all the cells in a healthy body. Among other objectives, the Human Cell Atlas would serve as a benchmark to study how cancers differ from healthy tissues at the spatial level.
That tumors have high heterogeneity was already evident from standard imaging techniques. Spatial omics differ from previous work by “providing high-resolution molecular insights and shedding light on intricate biological interactions,” said Mustafa. In cancer treatment, this could be the difference that allows researchers to take on drug-resistant cancers.
“You often need multiple omics modalities to see the true extent of the heterogeneity because each omics level, regardless of whether it is spatial or bulk or single cell, is only capturing a sub-fraction of the molecular heterogeneity within a cell,” said Alice Ly, director of Spatial Biology Applications at Aspect Analytics, a Belgian company that develops custom software solutions for spatial multiomics.
Despite a plethora of choices, the most common omics technologies in spatially resolved studies of tumors are transcriptomics and proteomics. Spatial transcriptomics and spatial proteomics are complementary. “If you find interesting profiles from the transcriptomics, you would use spatial proteomics to see which particular cellular phenotype has this or that transcriptional profile,” said Stadler.
As a growing field, spatial omics has yet to overcome many limitations. It is costly and requires diverse, technical expertise to bring together data from multiple high-throughput sequencing and imaging modalities. Analyzing this data is computationally expensive and time-consuming (files often run into hundreds of gigabytes). Furthermore, “while various pipelines and algorithmic programs have been developed to expedite and simplify data analysis, the interpretation of results remains a challenging and evolving facet,” explained Mustafa.
From Spatial Omics to Clinical Outcomes
Spatial omics describe the tumor landscape in extensive detail, but these insights need to translate into clinical outcomes to benefit cancer patients. Spatial omics are already generating clinically relevant insights. “Studies have shown that the immune system, the infiltration of different types of immune cells, has implications for how well a patient will respond to certain therapies,” said Stadler.
One way that spatial omics could be applied to clinical oncology is to look at many proven clinical markers in a tissue section to determine the specific tumor subtype. “It could also be used to transfer interesting findings from next-generation sequencing data of relevant markers to see whether they have a significant prognostic value,” added Stadler.
Despite its promises, computation remains a major bottleneck. Plummer stresses that integrating data from different technologies and analyzing them in a meaningful way is what's the field missing the most. “That is because the data sources are very large files and take days to run.”
Companies such as Aspect Analytics, Owkin, and Aspect Analytics are working to change that with artificial intelligence. Aspect Analytics's technology enables multimodal investigation of tissue sections. The company has registration strategies to “overlay data sets from different modalities, different vendors, different technologies and so forth, to create a common coordinate framework to allow combined analysis of these data sets,” says Ly.
Working with partners in industry and academia, Owkin is building MOSAIC, a spatial multiomics platform. By generating spatial omics data and analyzing it in combination with multimodal data of samples from thousands of patients using AI, it hopes to go further in understanding the molecular bases of different cancers.
For instance, the platform could enable accurate diagnosis of many cancers with tricky diagnoses, such as triple-negative breast cancer (TNBC). TNBC gets its name from its lack of the three receptors commonly found in breast cancers, a condition that makes diagnosing it difficult. “When someone gets TNBC, what they really have is one of 20 diseases or so and no one knows what they are. Most of the drugs that you might try on them are going to be the ones that aren't going to fit their biology,” said Lehar.
Regarding TNBC, Plummer said that “we have seen that even within those tumors that are age-matched and matched for TNBC, there are changes within the cell neighborhoods.” This presents a formidable challenge for MOSAIC and other spatial omics projects: to identify molecular subtypes of TNBC that reveal the different biologies happening for different patients.
In the more immediate term, Plummer sees spatial omics to be applied more broadly at St. Jude's. Many patients come there following unsuccessful standard-of-care treatments for pediatric cancer at other medical institutes. Plummer suggested that those are the cases where spatial oncology could have its first wins. “We want to show effectively, if we have clinical trials, all the levels of information by which we've changed that tumor, and that tumor is now getting attacked and necrotic and going away.”
Cancer biologists are excited about overlaying omics data over tissue images to generate multilayered pictures of a tumor. This allows them to produce a physical framework to map tumors in rich detail, learning about how differences along the spatial coordinates of a tumor shape its biology. As the field overcomes its technical limitations, it will open up clinical applications for both prognostics and treatment. By looking at tumors in rich 2D and 3D maps and at different stages, researchers will learn more about how tumors evolve in response to treatments.
ITALIANO
I ricercatori stanno utilizzando l’omica spaziale per osservare più in profondità il microambiente tumorale e svelare l’eterogeneità del tumore con l’obiettivo di raccogliere importanti informazioni cliniche.
Molti tumori difficili da trattare spesso recidivano mesi o anni dopo il successo del trattamento. "Potresti avere una cellula che sfugge al trattamento ed è quella cellula che si popolerà e sarà resistente e consentirà il verificarsi di una recidiva", ha affermato Jasmine Plummer, direttrice fondatrice del Center for Spatial Omics presso il St. Jude Children's Research Hospital.
Lo studio di tali tumori anche con tecnologie omiche a cellula singola potrebbe non individuare queste cellule resistenti. Prima dell’analisi, le tecnologie monocellulari distruggono il tessuto tumorale per osservare cosa sta accadendo nel tessuto nel suo insieme. Tuttavia, molti degli aspetti interessanti all’interno dei tumori accadono a livello delle singole cellule e dipendono dal contesto in cui esistono. Le tecnologie a cellula singola perdono questo contesto spaziale quando le cellule vengono disgregate.
È qui che entrano in gioco le omiche spaziali.
Con i progressi nelle tecnologie omiche, i biologi oncologici dispongono di ampie informazioni sui geni, sulle proteine e su altri metaboliti che costituiscono l’ambiente disordinato di un tumore. L'omica unicellulare va oltre, consentendo l'identificazione di tutti i tipi di cellule in un campione di tumore. Ciò ha solo approfondito la nostra comprensione dell’estrema eterogeneità delle cellule tumorali. Le tecnologie omiche spaziali stanno collocando queste intuizioni nel contesto spaziale.
Prendiamo ad esempio l'espressione genetica. La trascrittomica unicellulare rivela quali geni vengono espressi nei diversi tipi di cellule. Ma non dice dove si trovano queste cellule nel tumore. Le tecnologie di trascrittomica spaziale colmano questa lacuna registrando simultaneamente le coordinate spaziali con i dati di espressione genica. Questo è il punto cruciale del campo in crescita delle omiche spaziali: assegnare codici pin ai dati omici.
Le tecnologie di trascrittomica spaziale come l'ibridazione in situ ed il sequenziamento in situ consentono ai ricercatori di catturare trascrittomi senza perdere informazioni spaziali. Il primo utilizza sonde fluorescenti gene-specifiche che legano gli mRNA, mentre il secondo sequenzia le trascrizioni direttamente in una sezione di un tessuto fissato.
A complemento di questi metodi basati sull’imaging ci sono altre tecnologie spaziali basate sul sequenziamento di prossima generazione. Questi includono la trascrittomica spaziale ad alta definizione (HDST) ed i codici a barre deterministici nei tessuti per il sequenziamento omico spaziale (DBiT-Seq). L'HDST utilizza matrici di sfere con codice a barre spaziali per mappare l'RNA nella posizione sulle sezioni istologiche. DBiT fa lo stesso sia per le proteine che per l'RNA, consentendo di studiare le interazioni RNA-proteine con un contesto spaziale. Le tecnologie per studiare le proteine ed altri metaboliti sono simili in linea di principio a quelle sviluppate per studiare le trascrizioni. Più recentemente, la spinta verso l’investigazione di più livelli di informazioni contemporaneamente sta guidando lo sviluppo di strumenti multiomici spaziali come DBiT.
Il diverso microambiente tumorale
Le cellule tumorali devono continuamente eludere il sistema immunitario mentre creano l’infrastruttura per supportare la loro crescita incontrollata. Tale infrastruttura richiede un’attenta comunicazione con una cerchia diversificata. Comprende, tra gli altri, cellule sane nel tessuto ospite, cellule immunitarie che devono essere ingannate e vasi sanguigni di cui il tumore ha bisogno per diffondersi. Questi costituiscono un ecosistema dinamico noto come microambiente tumorale (TME) che svolge un ruolo fondamentale nella crescita e nella diffusione di un tumore.
Integrando l'omica unicellulare e l'omica spaziale, i ricercatori hanno finalmente un modo per creare mappe dettagliate di diversi tessuti, compresi i tumori. In un articolo pubblicato su Molecular Systems Biology, i ricercatori hanno creato un atlante unicellulare del fegato umano TME. I ricercatori hanno scoperto interazioni ricorrenti tra le cellule del carcinoma e le cellule stromali circostanti, suggerendo che i farmaci che prendono di mira queste interazioni potrebbero essere applicabili in modo più ampio.
In un altro studio pubblicato su Cell, gli scienziati hanno dimostrato che le cellule T immunitarie presentano una notevole diversità fenotipica nella TME del seno. Non solo superano di gran lunga i tipi di cellule T nei tessuti mammari sani, ma anche gli stati di attivazione dei tipi di cellule T tumorali sono in un continuum. Inoltre, lo studio ha suggerito che la diversità delle cellule immunitarie fosse una conseguenza della diversità dei microambienti locali all’interno del tumore.
I tumori hanno una complicata ecologia immunitaria che li aiuta a “diventare invisibili al sistema immunitario od a dire al sistema immunitario di smettere di annusare in modo da non trovarlo od a dire alle cellule T di non tentare di uccidere le cellule tumorali”, ha affermato Joseph Lehar, senior vicepresidente della strategia di ricerca e sviluppo presso Owkin, una startup franco-americana di biotecnologie basate sull'intelligenza artificiale.
Nella biologia immunitaria dei tumori, le decisioni prese dalle cellule sull’opportunità di uccidere le cellule sospettate di essere tumori sono altamente localizzate. "Tutto dipende dall'aspetto di una particolare cellula tumorale o di un gruppo di cellule e dalle cellule immunitarie che lo percepiscono e quindi rispondono in un modo particolare", ha aggiunto Lehar. Una comprensione spaziale del microambiente immunitario dei tumori è fondamentale per poter affrontare i tumori incurabili. Questo perché la capacità di un tumore di eludere il sistema immunitario è fondamentale sia per il modo in cui stabilisce l’architettura necessaria per crescere sia per il modo in cui resiste ai trattamenti.
Un altro aspetto critico della TME è la presenza di una nicchia ipossica. Un fenomeno che caratterizza i tumori maligni, si tratta di un livello di ossigeno inferiore a quello fisiologico che è alla base della diversa architettura di segnalazione cellulare all'interno dei tumori. In uno studio pubblicato su Immunity, i ricercatori hanno dimostrato che le nicchie ipossiche attraggono e nascondono le cellule immunitarie tumorali. Combinando le informazioni spaziali con la trascrittomica di una singola cellula, hanno dimostrato che la diafonia tra il glioblastoma e le cellule immunitarie nella nicchia ipossica è fondamentale per sopprimere il sistema immunitario.
La TME è costituita anche da ulteriori meccanismi che determinano il destino di un tumore attraverso le loro interazioni sia con il cancro che con le cellule sane. Questi includono la matrice extracellulare, i lipidi liberi ed i segnali meccanici, solo per citarne alcuni. Per mappare questi componenti lungo il tumore, i ricercatori sfruttano altre tecnologie di profilazione spaziale, spesso utilizzate insieme all’omica spaziale, come l’imaging multiplex e la citometria di massa.
Nell’imaging multiplex, “usiamo anticorpi per rilevare una vasta gamma di proteine diverse nella stessa sezione di tessuto”, ha affermato Charlotte Stadler, codirettrice della piattaforma di biologia spaziale e unicellulare e capo dell’unità di proteomica spaziale presso il centro di ricerca nazionale svedese SciLifeLab. Ciò consente ai ricercatori di eseguire “una fenotipizzazione approfondita e di sapere quali tipi di cellule sono vicini nello spazio e potenzialmente interagiscono tra loro”. La fenotipizzazione profonda, un termine sempre più focalizzato nello spazio della medicina di precisione, si riferisce alla fenotipizzazione a più livelli di dati omici.
La citometria di massa, allo stesso modo, consente ai ricercatori di tracciare molti più metaboliti rispetto alle tecniche di citometria convenzionali. Tali tecniche di profilazione spaziale sono fondamentali anche per consentire omiche spaziali tridimensionali (3D). Mentre le mappe dei tumori ottenute con l'omica spaziale sono spesso bidimensionali (2D), i tumori stessi esistono in 3D. Le omiche spaziali 3D forniscono un quadro completo di ciò che sta accadendo all’interno dei tumori e, di conseguenza, informazioni migliori.
I tumori sono altamente eterogenei
Cellule diverse all'interno di un tumore hanno una diversa composizione genotipica e fenotipica. Anche in una popolazione geneticamente omozigote di cellule tumorali all'interno di un tumore, le cellule differiscono considerevolmente dal punto di vista fenotipico. L'instabilità genomica, considerata un importante segno distintivo dello sviluppo del tumore, è il principale fattore dell'elevata diversità cellulare nei tumori.
L’elevata diversità delle cellule all’interno di un tumore “è fondamentale per comprendere le molteplici funzioni delle cellule tumorali e la loro intricata relazione con il microambiente”, ha affermato Dana Adel Mustafa, ricercatrice sul cancro presso l’Erasmus University Medical Center.
Applicando tecniche di omica spaziale ai tessuti tumorali, i biologi stanno imparando di più sull’eterogeneità dei diversi tumori. Le tecnologie omiche spaziali stanno facendo rapidi progressi nella ricerca sul cancro producendo dati che generano ipotesi sull’eterogeneità del tumore. Ad esempio, in uno studio pubblicato su Cancer Cell, i ricercatori hanno utilizzato la trascrittomica spaziale per studiare l’eterogeneità nel carcinoma a cellule renali. Hanno osservato che l’eterogeneità intratumorale supera di gran lunga quella delle mutazioni somatiche nel cancro del rene.
Più specificamente, la posizione delle cellule T immunitarie nel tessuto, piuttosto che le mutazioni che hanno accumulato, definisce principalmente il livello della loro disfunzione. Questo studio faceva parte del consorzio Human Cell Atlas, uno sforzo più ampio per creare un atlante completo di tutte le cellule di un corpo sano. Tra gli altri obiettivi, l’Atlante delle cellule umane servirebbe da punto di riferimento per studiare in che modo i tumori differiscono dai tessuti sani a livello spaziale.
Che i tumori abbiano un’elevata eterogeneità era già evidente dalle tecniche di imaging standard. Le omiche spaziali differiscono dal lavoro precedente perché “forniscono intuizioni molecolari ad alta risoluzione e fanno luce su complesse interazioni biologiche”, ha affermato Mustafa. Nel trattamento del cancro, questa potrebbe essere la differenza che consente ai ricercatori di affrontare i tumori resistenti ai farmaci.
"Spesso sono necessarie più modalità omiche per vedere la reale portata dell'eterogeneità perché ogni livello omico, indipendentemente dal fatto che sia spaziale, di massa o di singola cellula, cattura solo una sottofrazione dell'eterogeneità molecolare all'interno di una cellula", ha affermato Alice. Ly, direttore delle applicazioni di biologia spaziale presso Aspect Analytics, una società belga che sviluppa soluzioni software personalizzate per la multiomica spaziale.
Nonostante una pletora di scelte, le tecnologie omiche più comuni negli studi sui tumori risolti spazialmente sono la trascrittomica e la proteomica. La trascrittomica spaziale e la proteomica spaziale sono complementari. "Se trovi profili interessanti dalla trascrittomica, utilizzeresti la proteomica spaziale per vedere quale particolare fenotipo cellulare ha questo o quel profilo trascrizionale", ha detto Stadler.
Essendo un campo in crescita, l’omica spaziale deve ancora superare molti limiti. È costoso e richiede competenze tecniche diverse per riunire dati provenienti da più modalità di sequenziamento e imaging ad alto rendimento. L'analisi di questi dati è computazionalmente costosa e richiede molto tempo (i file spesso occupano centinaia di gigabyte). Inoltre, “sebbene siano stati sviluppati vari processi e programmi algoritmici per accelerare e semplificare l’analisi dei dati, l’interpretazione dei risultati rimane un aspetto impegnativo e in evoluzione”, ha spiegato Mustafa.
Dalle omiche spaziali ai risultati clinici
Le omiche spaziali descrivono il panorama tumorale in modo molto dettagliato, ma queste intuizioni devono tradursi in risultati clinici a beneficio dei pazienti affetti da cancro. Le omiche spaziali stanno già generando intuizioni clinicamente rilevanti. "Gli studi hanno dimostrato che il sistema immunitario, l'infiltrazione di diversi tipi di cellule immunitarie, ha implicazioni sulla risposta di un paziente a determinate terapie", ha affermato Stadler.
Un modo in cui l’omica spaziale potrebbe essere applicata all’oncologia clinica è quello di esaminare molti marcatori clinici comprovati in una sezione di tessuto per determinare il sottotipo specifico di tumore. "Potrebbe anche essere utilizzato per trasferire risultati interessanti dai dati di sequenziamento di prossima generazione di marcatori rilevanti per vedere se hanno un valore prognostico significativo", ha aggiunto Stadler.
Nonostante le sue promesse, il calcolo rimane un grosso collo di bottiglia. Plummer sottolinea che integrare dati provenienti da diverse tecnologie e analizzarli in modo significativo è ciò che manca di più in questo campo. "Questo perché le origini dati sono file molto grandi e richiedono giorni per essere eseguite."
Aziende come Aspect Analytics, Owkin e Aspect Analytics stanno lavorando per cambiare la situazione con l’intelligenza artificiale. La tecnologia di Aspect Analytics consente l'indagine multimodale di sezioni di tessuto. L’azienda dispone di strategie di registrazione per “sovrapporre set di dati provenienti da diverse modalità, diversi fornitori, diverse tecnologie e così via, per creare un quadro di coordinate comune per consentire l’analisi combinata di questi set di dati”, afferma Ly.
Lavorando con partner dell’industria e del mondo accademico, Owkin sta costruendo MOSAIC, una piattaforma multiomica spaziale. Generando dati omici spaziali e analizzandoli in combinazione con dati multimodali di campioni di migliaia di pazienti che utilizzano l’intelligenza artificiale, si spera di andare oltre nella comprensione delle basi molecolari di diversi tumori.
Ad esempio, la piattaforma potrebbe consentire una diagnosi accurata di molti tumori con diagnosi complicate, come il cancro al seno triplo negativo (TNBC). Il TNBC prende il nome dalla mancanza dei tre recettori comunemente presenti nel cancro al seno, una condizione che ne rende difficile la diagnosi. “Quando qualcuno si ammala di TNBC, quello che ha realmente è una delle 20 malattie circa e nessuno sa cosa siano. La maggior parte dei farmaci che potresti provare su di loro saranno quelli che non si adatteranno alla loro biologia", ha detto Lehar.
Per quanto riguarda il TNBC, Plummer ha affermato che "abbiamo visto che anche all'interno di quei tumori che sono abbinati per età e abbinati per TNBC, ci sono cambiamenti nelle vicinanze delle cellule". Ciò rappresenta una sfida formidabile per MOSAIC e altri progetti di omica spaziale: identificare sottotipi molecolari di TNBC che rivelino le diverse biologie che si verificano per i diversi pazienti.
In termini più immediati, Plummer vede che l'omica spaziale verrà applicata in modo più ampio a St. Jude. Molti pazienti arrivano lì dopo aver ricevuto insuccessi trattamenti standard per il cancro pediatrico presso altri istituti medici. Plummer ha suggerito che questi sono i casi in cui l’oncologia spaziale potrebbe ottenere le prime vittorie. "Vogliamo mostrare in modo efficace, se avremo studi clinici, tutti i livelli di informazione con cui abbiamo cambiato quel tumore, e quel tumore ora viene attaccato, diventa necrotico e scompare."
I biologi oncologici sono entusiasti di sovrapporre i dati omici alle immagini dei tessuti per generare immagini multistrato di un tumore. Ciò consente loro di produrre una struttura fisica per mappare i tumori in modo dettagliato, apprendendo come le differenze lungo le coordinate spaziali di un tumore modellano la sua biologia. Man mano che il campo supererà i suoi limiti tecnici, aprirà applicazioni cliniche sia per la prognosi che per il trattamento. Osservando i tumori in ricche mappe 2D e 3D ed in diverse fasi, i ricercatori impareranno di più su come i tumori si evolvono in risposta ai trattamenti.
Da:
https://www.liebertpub.com/doi/10.1089/genbio.2023.29115.sro?utm_campaign=Resend%20of%20GENBIO%20FP%20Oct%2026%202023&utm_content=Article-2&utm_term=&utm_medium=email&utm_source=Adestra



Commenti
Posta un commento